6.5 KiB
6.5 KiB
FScanpy
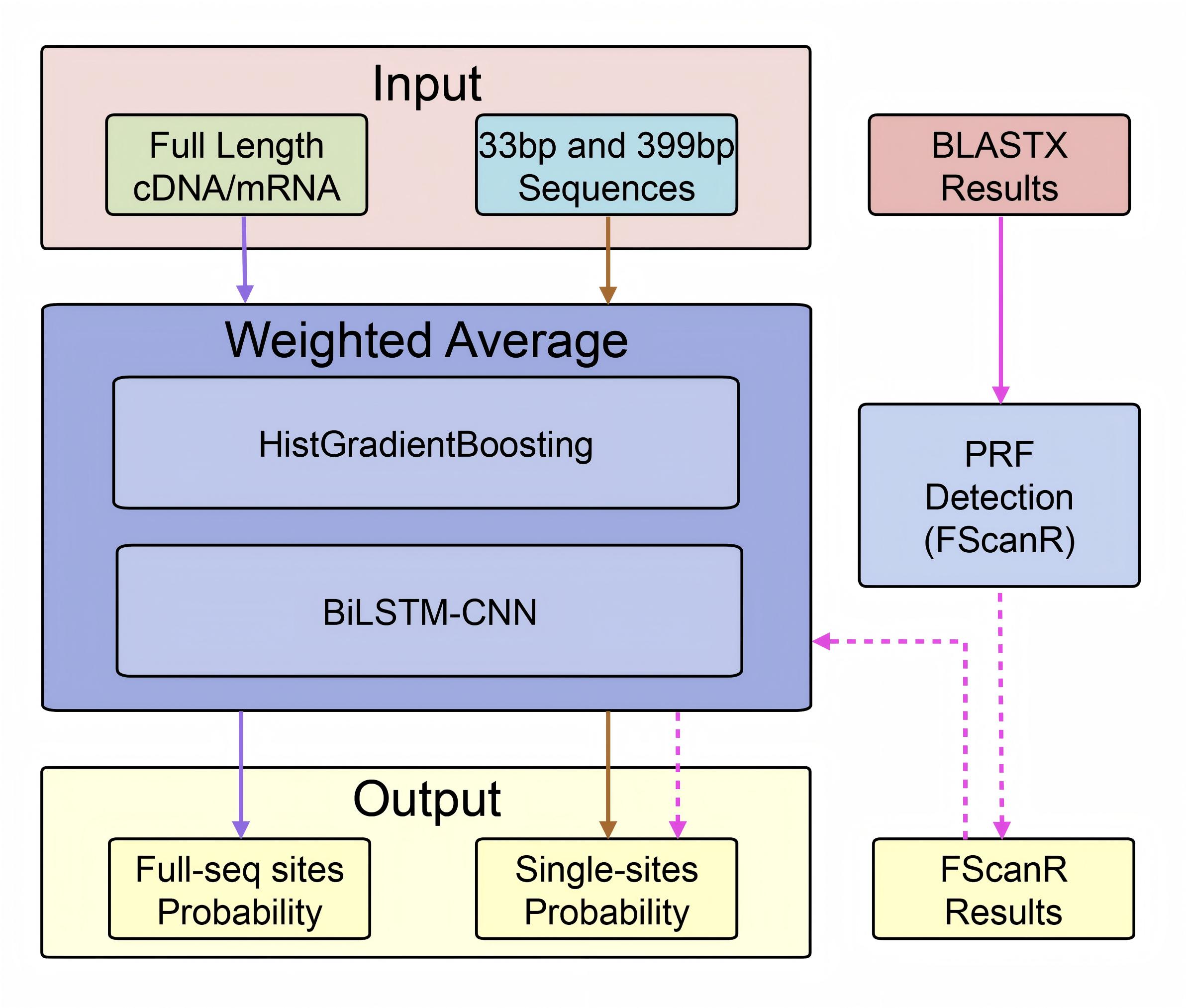
基于机器学习的程序性核糖体移码预测框架
FScanpy 是一个专为预测核苷酸序列中程序性核糖体移码 (PRF) 位点而设计的综合性 Python 包。通过将先进的机器学习方法(HistGradientBoosting 和 BiLSTM-CNN)与已建立的 FScanR 框架相结合,FScanpy 提供了稳健且准确的 PRF 位点预测。
🔧 安装
前置条件
- Python ≥ 3.9
- 所有依赖项将自动安装
通过 pip 安装(推荐)
pip install FScanpy
从源码安装
git clone https://github.com/your-org/FScanpy-package.git
cd FScanpy-package
pip install -e .
🚀 快速开始
基本用法
from FScanpy import predict_prf
# 简单序列预测
sequence = "ATGCGTACGTTAGC..." # 您的 DNA 序列
results = predict_prf(sequence=sequence)
# 查看前十个预测结果
print(results[['Position', 'Ensemble_Probability', 'Short_Probability', 'Long_Probability']].head(10))
可视化
from FScanpy import plot_prf_prediction
# 生成预测图表
results, fig = plot_prf_prediction(
sequence=sequence,
short_threshold=0.65, # HistGB 阈值
long_threshold=0.8, # BiLSTM-CNN 阈值
ensemble_weight=0.4, # 40% 短模型,60% 长模型
title="PRF 预测结果"
)
高级用法
from FScanpy import PRFPredictor
import pandas as pd
# 创建预测器实例
predictor = PRFPredictor()
# 对预提取区域进行批量预测
data = pd.DataFrame({
'Long_Sequence': ['ATGCGT...' * 60, 'GCTATAG...' * 57] # 399bp 序列
})
results = predictor.predict_regions(data, ensemble_weight=0.4)
# 使用自定义参数进行序列级预测
results = predictor.predict_sequence(
sequence=sequence,
window_size=1, # 滑动窗口步长
ensemble_weight=0.3, # 模型权重
short_threshold=0.5 # 过滤阈值
)
🎛️ 集成权重配置
ensemble_weight 参数控制 HistGB 和 BiLSTM-CNN 模型的权重比例:
| ensemble_weight | HistGB 模型 | BiLSTM-CNN 模型 | 特性 | 最适用于 |
|---|---|---|---|---|
| 0.2-0.3 | 20-30% | 70-80% | 高特异性,减少假阳性 | 精确验证、临床应用 |
| 0.4 | 40% | 60% | 最优平衡,最高 AUC | 标准分析(推荐) |
| 0.6-0.8 | 60-80% | 20-40% | 高敏感性,捕获更多位点 | 高通量筛选、探索研究 |
模型特性说明
- HistGB 模型:擅长识别真阴性样本,预测保守,假阳性率低
- BiLSTM-CNN 模型:擅长识别真阳性样本,预测敏感,能捕获更多潜在位点
权重选择示例
# 高特异性配置(偏向 HistGB)
precise_results = predict_prf(sequence, ensemble_weight=0.25)
# 最优平衡配置(4:6 比例)
balanced_results = predict_prf(sequence, ensemble_weight=0.4)
# 高敏感性配置(偏向 BiLSTM-CNN)
sensitive_results = predict_prf(sequence, ensemble_weight=0.7)
📊 核心函数
主要预测接口
predict_prf(
sequence=None, # 单/多序列或 None
data=None, # 包含 399bp 序列的 DataFrame 或 None
window_size=3, # 滑动窗口步长
short_threshold=0.1, # 短模型过滤阈值
ensemble_weight=0.4, # 短模型权重 (0.0-1.0)
model_dir=None # 自定义模型目录
)
可视化函数
plot_prf_prediction(
sequence, # 输入 DNA 序列
window_size=3, # 扫描步长
short_threshold=0.65, # 短模型绘图阈值
long_threshold=0.8, # 长模型绘图阈值
ensemble_weight=0.4, # 模型权重
title=None, # 图表标题
save_path=None, # 保存文件路径
figsize=(12,8), # 图形大小
dpi=300 # 保存图表的分辨率
)
PRFPredictor 类方法
predictor = PRFPredictor()
# 序列预测(滑动窗口)
predictor.predict_sequence(sequence, ensemble_weight=0.4)
# 区域预测(批处理)
predictor.predict_regions(dataframe, ensemble_weight=0.4)
# 特征提取
predictor.extract_features(sequences)
# 模型信息
predictor.get_model_info()
📈 输出字段
预测结果
Position:在原始序列中的位置Ensemble_Probability:最终集成预测(主要结果)Short_Probability:HistGradientBoosting 预测 (0-1)Long_Probability:BiLSTM-CNN 预测 (0-1)Ensemble_Weights:使用的模型权重配置
序列信息
Short_Sequence:短模型使用的 33bp 序列Long_Sequence:长模型使用的 399bp 序列Codon:预测位置的 3bp 密码子Sequence_ID:多序列输入的标识符
🔬 与 FScanR 集成
FScanpy 与 FScanR 流程无缝协作,提供全面的 PRF 分析:
from FScanpy import fscanr, extract_prf_regions, predict_prf
# 步骤 1:使用 FScanR 进行 BLASTX 分析
blastx_results = fscanr(
blastx_data,
mismatch_cutoff=10,
evalue_cutoff=1e-5,
frameDist_cutoff=10
)
# 步骤 2:提取 PRF 候选区域
prf_regions = extract_prf_regions(original_sequence, blastx_results)
# 步骤 3:使用 FScanpy 进行预测
final_predictions = predict_prf(data=prf_regions, ensemble_weight=0.4)
📚 文档
📝 引用
如果您在研究中使用 FScanpy,请引用:
@software{fscanpy2024,
title={FScanpy: A Machine Learning Framework for Programmed Ribosomal Frameshifting Prediction},
author={[作者姓名]},
year={2024},
url={https://github.com/your-org/FScanpy}
}
🏗️ 依赖项
FScanpy 会自动安装所有必需的依赖项:
numpy>=1.24.3pandas>=2.2.3tensorflow>=2.10.1scikit-learn>=1.6.0matplotlib>=3.9.4joblib>=1.4.2biopython>=1.85wrapt>=1.17.0
FScanpy - 通过机器学习推进程序性核糖体移码研究 🧬